nat microbiol|全球肠道微生物群揭示肠杆菌科的双面性:是威胁还是共生?

英文标题:Ecological dynamics of Enterobacteriaceae in the human gut microbiome across global populations
中文标题:全球人群肠道微生物群中肠杆菌科的生态动态
发表期刊:nature microbiology
影响因子:20.5
研究背景
人体肠道微生物群对健康意义重大,其失衡与多种疾病相关。肠杆菌科细菌是常见的机会致病菌,在健康人肠道中也普遍存在,提示种间相互作用可能影响感染抗性。但现有对肠杆菌科的研究多聚焦临床分离株,且以往探究其与肠道微生物群关系的研究受样本量和技术限制。如今宏基因组学发展提供了新契机,本研究利用大规模宏基因组数据,旨在揭示全球人群肠道微生物群中肠杆菌科的生态动态,为相关疾病防治和非抗生素疗法开发提供依据。
研究结果
1、肠杆菌科的全球分布
为全面表征与肠杆菌科定植相关的人类肠道微生物群特征,研究者从45个国家的(欧洲35%、北美27.5%、亚洲23.2%、非洲8.4%)65项研究中检索了12,238份公开的人类肠道宏基因组样本(图1)。基于统一人类胃肠道基因组(UHGG)目录,准确检测了全球宏基因组中4,612种肠道微生物(113种属于肠杆菌科)的存在和丰度。
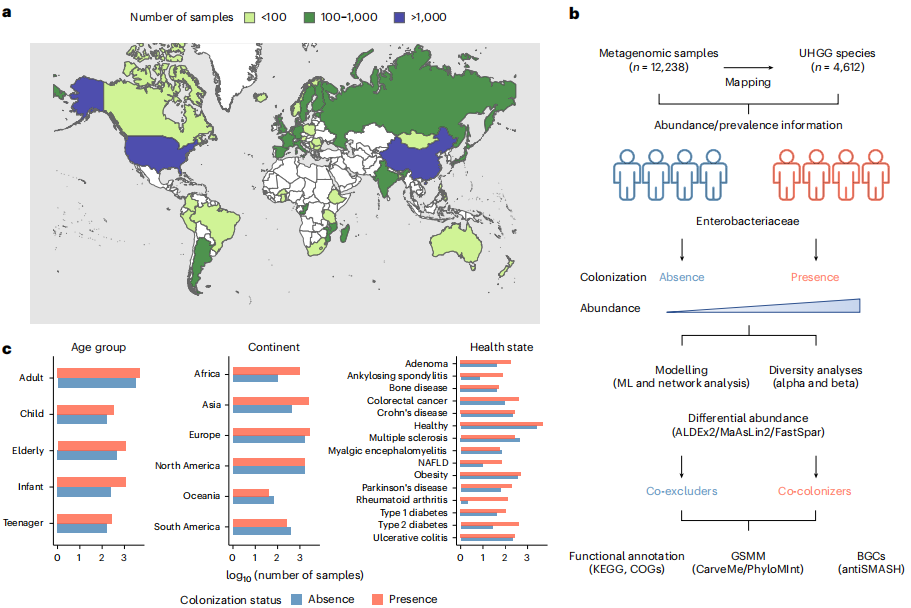
图1. 探索肠杆菌科的全球生态景观
研究发现,肠杆菌科的总体患病率为66%,其分布受地理、宿主状态显著影响:非洲样本中大肠杆菌患病率最高(88%),婴儿(74%)及类风湿性关节炎患者(96%)显著富集(图2a)。研究者进一步分析了肠杆菌科物种在肠道宏基因组中的共分布模式(图2b),发现亚洲样本中大肠杆菌与肺炎克雷伯菌共定植率达16.2%(P<0.001)。此外,在非洲和大洋洲样本中,大肠杆菌、肺炎克雷伯菌和霍氏肠杆菌显著共现(观察比例5.3%vs预期比例0.7%)。总体而言,这些肠杆菌科共定植的地理差异可能反映了环境条件、饮食习惯、生活方式和医疗实践的差异。研究通过宏基因组多位点序列分型(MLST)分析5,128份健康成人样本,发现大肠杆菌存在585种序列型(STs),其中76.5%为未知STs(如非洲高丰度的 ST100024/83),揭示全球未被充分研究的菌株多样性。
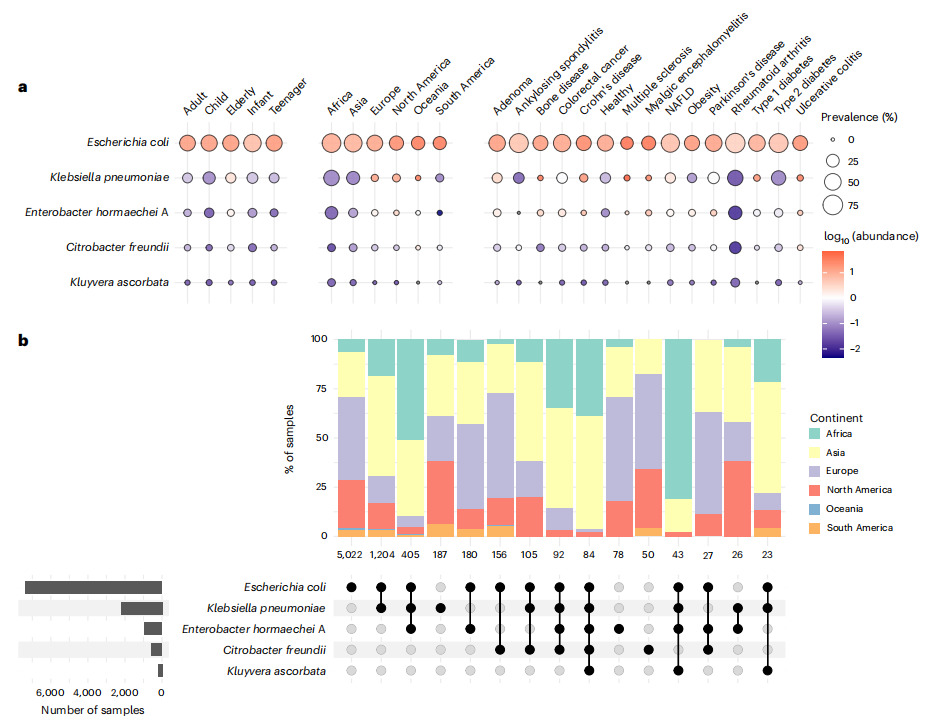
图2. 最常见肠杆菌科物种的分布与多样性
2、肠道微生物群结构与肠杆菌科定植动态的关联
利用全球12,000份肠道宏基因组数据,研究者探索微生物群组成与肠杆菌科定植状态的关系。基于非肠杆菌科物种的丰度和患病率,构建机器学习分类器来区分样本是否含肠杆菌科(图3a)。以肠杆菌科整体或大肠杆菌、肺炎克雷伯菌的定植状态为因变量,测试三种监督学习方法,梯度提升法表现最优。
多样性分析显示,肠杆菌科阳性样本的β多样性显著高于阴性样本,且与α多样性和测序深度无关。差异丰度分析结合ALDEx2和MaAsLin2模型,控制多种因素后,在众多样本中鉴定出与肠杆菌科等显著相关的物种,分为共定植菌和共排斥菌(图3b)。分类学特征显示,毛螺菌目(Lachnospirales)、颤螺旋菌目(Oscillospirales)和拟杆菌目(Bacteroidales)多为共排斥菌,乳杆菌目(Lactobacillales)、韦荣球菌目(Veillonellales)和放线菌目(Actinomycetales)与共定植菌相关(图3c)。
临床相关性验证结果表明,本研究中所鉴定出的共排斥菌/共定植菌,与针对产碳青霉烯酶肠杆菌科(CPE)的队列研究中发现的差异物种之间存在显著重叠。这一现象提示,这些物种或许并非仅对肠杆菌科的整体定植产生影响,而是有可能特异性地作用于CPE的定植过程。
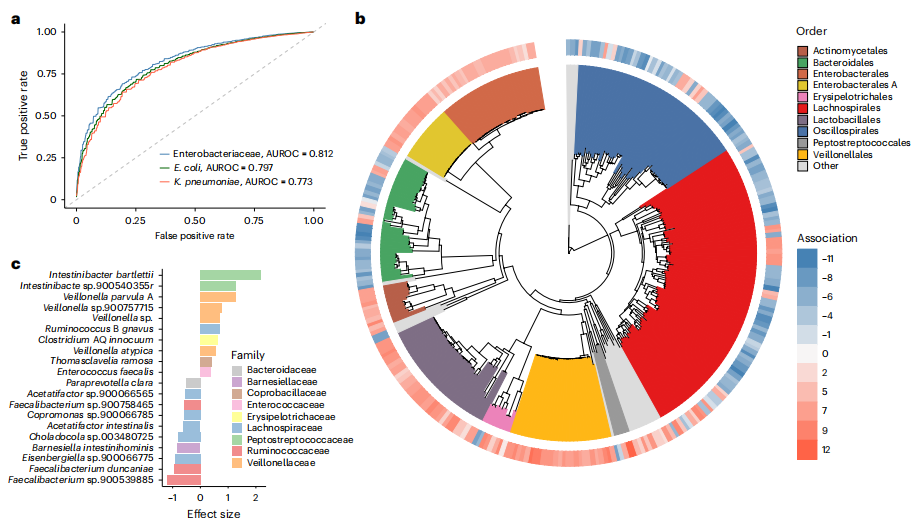
图3. 肠道微生物组组成与肠杆菌科的定植和丰度有关
3、肠杆菌科定植的菌株特异性模式
上述分析揭示微生物组物种组成与肠杆菌科定植模式联系紧密,然而菌株水平的差异也可能与肠杆菌科-微生物组之间的相互作用有关,研究者针对39种在健康成年人肠道内,与肠杆菌科存在共定植或共排斥关系的微生物展开研究。这些物种在统一人类胃肠道基因组(UHGG)中有充足高质量基因组代表。
借助统一人类胃肠道蛋白质(UHGP)目录,研究者分析了这39个物种的附属基因组,识别与肠杆菌科定植相关的亚种群体。结果发现部分附属基因与肠杆菌科定植状态显著相关,且多富集于核苷酸转运和代谢相关功能,主要涉及多形瘤胃球菌(Ruminococcus B gnavus)和福氏粪单胞菌(Faecalimonas phoceensis)。
研究者进一步研究了与肠杆菌科相关的附属基因数量最多的菌株(前10%的菌株)的系统发育相似性。结果表明,与Ruminococcus B gnavus基因组相比,Faecalimonas phoceensis中与肠杆菌科共定植相关的种群结构要强得多(图4)。在众多候选物种中,只有2种粪单胞菌属物种被确定为肠杆菌科的共定植物种。相较于其他相关菌属,粪单胞菌属的多样性与肠杆菌科共定植之间存在更特定的菌株水平关联。
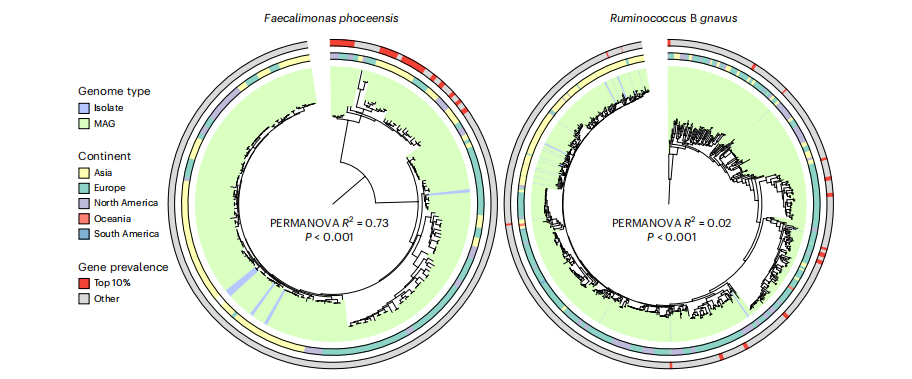
图4. 福氏粪单胞菌呈现出菌株特异性的共定植模式
4、共定植物种功能多样
作为分类学研究的补充,研究者分析307种物种中245种非肠杆菌科物种的功能和多样性,这些物种在不同数据集和分类群中呈共定植或共排斥模式。基于KEGG注释发现,共定植物种功能多样性更高(图5a),与注释覆盖范围和基因组质量无关,与更高的代谢独立性相关。统计分析KEGG直系同源基因(KOs)发现,共定植与耐药性、DNA调控功能关联强,共排斥与铁代谢、转运基因关联强,暗示肠杆菌科和共排斥物种存在铁竞争。研究者对与肠杆菌科共定植和共排斥相关的主要功能类别(直系同源聚类组,COGs)进行了特征描述(图5b),共定植物种富含代谢相关功能(如氨基酸、核苷酸和无机离子代谢),支持其代谢独立性更强的假设;共排斥物种编码更多信号转导相关基因(如孢子形成、运动性和群体感应),还有较多功能未知基因,或在抵抗定植中起作用,即使仅考虑芽孢杆菌门的物种(该门包含数量最多的共排斥物种和共定植物种),这些结果依然一致。利用gutSMASH算法研究发现,共排斥物种富含产短链脂肪酸及涉及 Rnf 复合物等的代谢基因簇(图5c)。这强化了粪杆菌属(肠道中显著的短链脂肪酸生产者之一)的相关信号,也凸显铁在共排斥物种中的重要性。综上,与肠杆菌科共存的物种功能多样,铁代谢和短链脂肪酸在调节肠道环境、调控肠杆菌科定植和丰度上或起重要作用。
研究者构建了候选共定植物种、共排斥物种及特定肠杆菌科物种的基因组规模代谢模型,计算代谢竞争和互补指数。结果显示,竞争与互补指数呈负相关,共定植物种与肠杆菌科的代谢距离低于共排斥物(图5d),表明生境筛选可能是肠杆菌科相关物种定植及微生物组组装的主因。此外,组内代谢距离小于组间,且模拟不同饮食培养基时结果一致,佐证了营养需求相似的肠道微生物易在个体间共存的观点 。
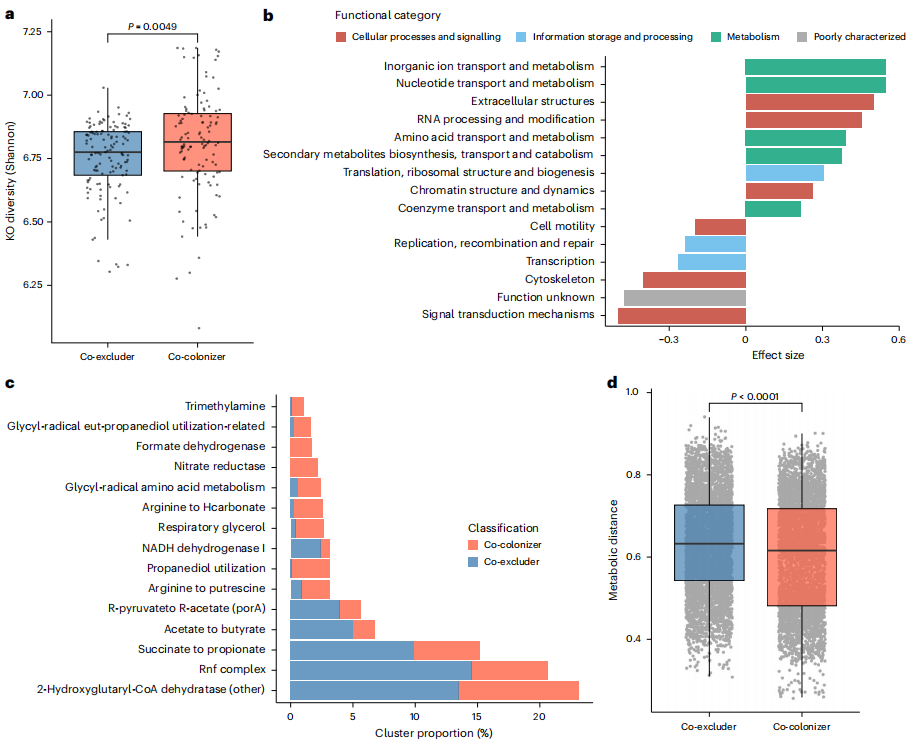
图5. 共排斥物种和共定植物种之间的功能差异
5、共排斥物种编码参与群体感应的基因
鉴于次级代谢产物影响细菌适存度与种间生态互作,研究者借助antiSMASH预测工具,分析肠杆菌科共定植物种和共排斥物种的生物合成基因簇(BGCs)。结果发现,共排斥物种显著富集环状内酯自体诱导物相关BGCs(图6a)。自体诱导物作为群体感应信号分子,与COG分析中,共排斥物种信号转导机制基因富集的结论相符。多数自体诱导物BGCs由毛螺菌科物种携带。按特定基因相似度分组(覆盖度超过50%且核苷酸同一性超过50%),识别出独特BGC家族,且这些家族在共定植物种和共排斥物种间存在差异(图6b)。将家族基因与 “生物合成基因簇最低限度信息” 数据库对比,发现与已知BGCs氨基酸同一性低(图6c),表明它们多未被表征。因群体感应分子能助力肠道微生物抵御外来病原体定植,共排斥物种中自体诱导物BGCs的富集,可能调控肠杆菌科在人体肠道的定植和丰度。
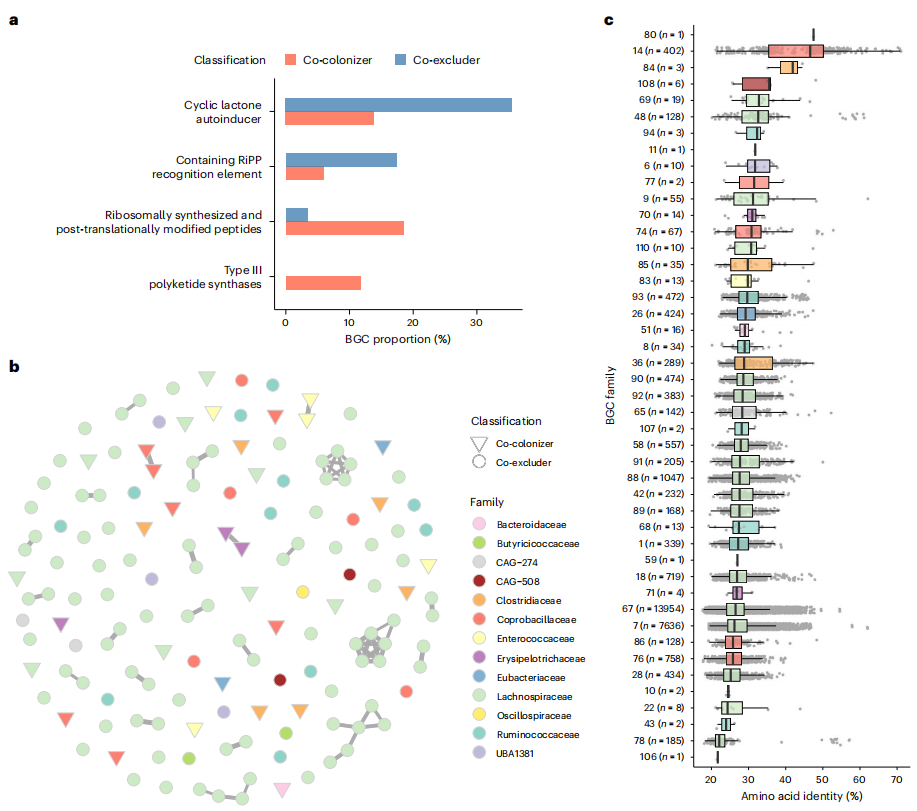
图6. 共排斥物种拥有参与群体感应的生物合成基因簇
研究小结
本研究聚焦肠杆菌科物种与肠道微生物组其他成员间的关系,综合采用多种研究方法,取得一系列重要发现。通过构建基因组规模代谢模型,深入分析代谢竞争和互补,揭示了生境筛选在肠道微生物定植和群落组装中的关键作用。进一步的代谢分析表明,共定植物种与共排斥物种在代谢功能上存在显著差异,这可能反映它们对肠道不同生态位的适应性差异。对生物合成基因簇的研究显示,共排斥物种中与群体感应相关的基因簇显著富集,这些基因簇或许在调控肠杆菌科的肠道定植和丰度方面发挥着重要作用。本研究为肠道微生物组的生态机制提供了新见解,有望为干预肠道微生物组以防治相关疾病提供理论支撑。
END
Peng 撰文
Winly 校稿
